FOREVER FISH
XTRA-ARTIKEL AUSGABE 2/2024
Die Fluoreszenz-in-situ-Hybridisierung (FISH) ist eine molekularzytogenetische Untersuchung mit langer Historie. Über ihren klinischen Nutzen sowie aktuelle Entwicklungen in der genetischen Diagnostik – der Humangenetiker PROF. THOMAS LIEHR im Interview
Herr Professor Liehr, warum ist die FISH-Diagnostik seinerzeit entwickelt worden?
Rückblickend sehe ich drei Gründe. Erstens wollte man die Bänderungszytogenetik weiter nutzen, zweitens aber ihre relativ geringe Auflösung überwinden und drittens eine Möglichkeit schaffen, auch Interphase-Zellen zu analysieren. Ab den 90ern gab es zunehmend Anbieter kommerzieller Sonden, und die diagnostische Anwendung explodierte. Das erste große Feld war die tumorgenetische Diagnostik an Meta- und Interphase-Chromosomen. Kommerzielle FISH-Sonden für die vergleichsweise seltenen angeborenen genetischen Erkrankungen standen erst um das Jahr 2000 bereit.
Wie kam es, dass Sie sich auf FISH-Diagnostik spezialisiert haben?
Als ich Anfang der 90er ein Diplomthema suchte, landete ich in der Humangenetik in Erlangen, wo Professor Gebhart gerade die FISH-Technik etablierte. Er und Professor Pfeiffer setzen mich an die FISH-Charakterisierung von zwölf Fällen, vor allem mit Katzenaugen-Syndrom, mit kleinen überzähligen Marker-Chromosomen. 1998 wechselte ich zu Professor Claussen nach Jena, wo seit Beginn der 90er eigene FISH-Sonden hergestellt wurden. Meine ehemalige Doktorandin Anja Weise hatte die Idee, eine große BAC-Sonden-Sammlung anzulegen. Somit haben wir seit etwa 2005 mehr als 8.000 FISH-Sonden zur Verfügung.
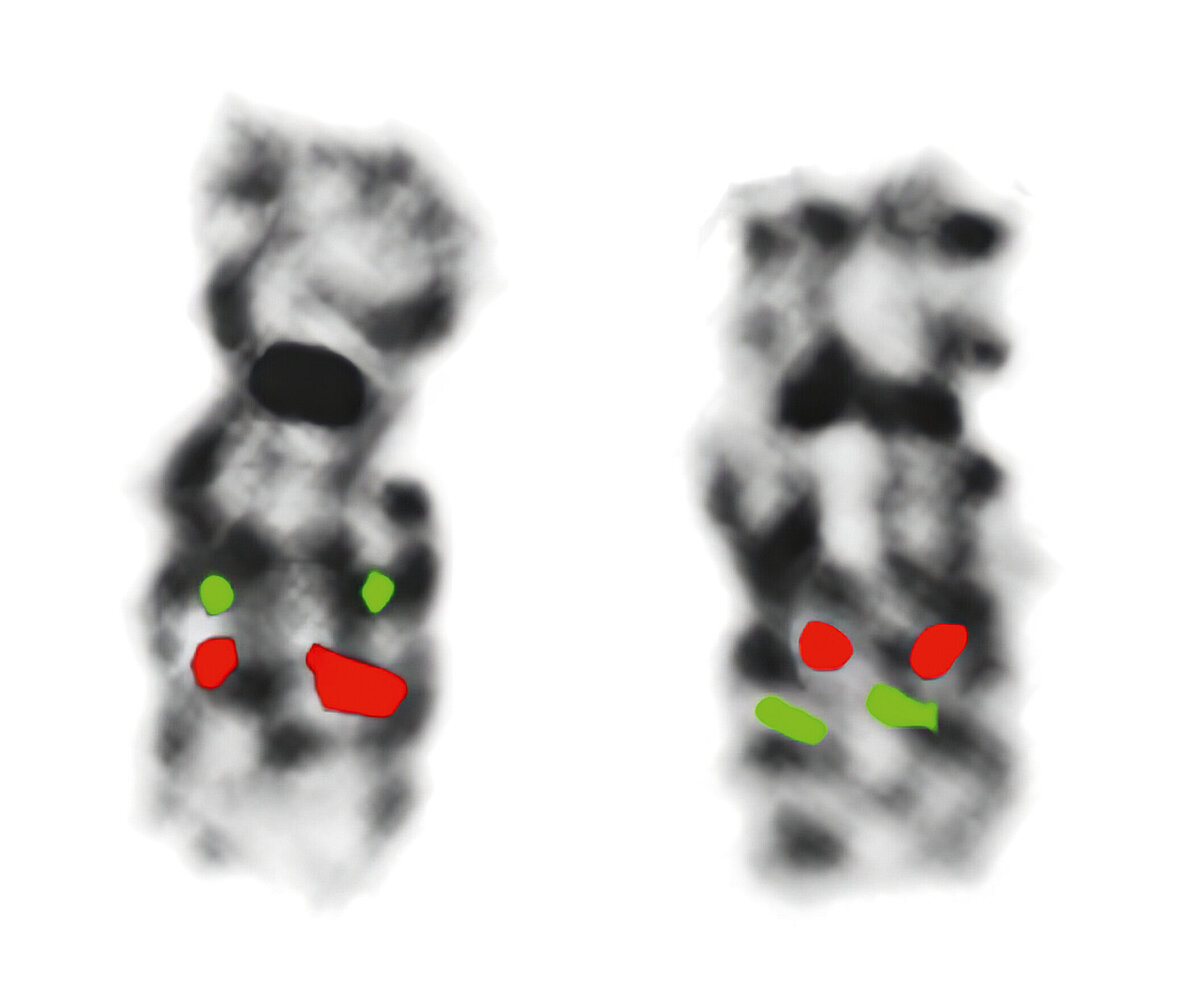
Was begeistert Sie an der FISH?
Die Möglichkeit, auf mikroskopischer Ebene genetische Veränderungen nachweisen, erkennen und klassifizieren zu können. Zudem ist die humangenetische Diagnostik extrem nah am Puls der Forschung. Wir bearbeiten seltene klinische Fälle, über die es meist nur wenige Publikationen gibt.
Wie hat sich die Humangenetik im Lauf der Zeit verändert?
Während Licht- und Fluoreszenzmikroskopie das Feld bis 2000 prägten, zeichnet sich nun eine wahrscheinlich unumkehrbare Entwicklung ab. Ging es bisher um Chromosomen, ihre Struktur und die genetische Beratung von Patienten, will man jetzt, mittels Hochdurchsatzmethoden und Bioinformatik, am liebsten „die eine Technik“ finden, mit der man alle genetischen Fragestellungen auf einmal lösen kann.
Sie klingen nicht begeistert?
Stimmt. Die frühere Blickdiagnostik erfahrener klinischer Genetiker soll durch Gesichtserkennungsprogramme für Krankheitsbilder oder durch blindes Screenen auf Veränderungen im Patientengenom ersetzt werden. Und das, ohne eine Verdachtsdiagnose zu benötigen. Zytogenetik und FISH werden als veraltete Methoden belächelt, obwohl Hunderttausende solcher Analysen jeden Monat allein in Deutschland durchgeführt werden.
Ohne FISH geht es also nicht?
Nein. Unser Labor in Jena bekommt, neben Einsendungen aus der eigenen Beratungsabteilung, mehrere Hundert Proben jährlich zur Abklärung von in Array-CGH oder Zytogenetik auffälligen Befunden. Die Zahlen steigen in den vergangenen Jahren eher an.
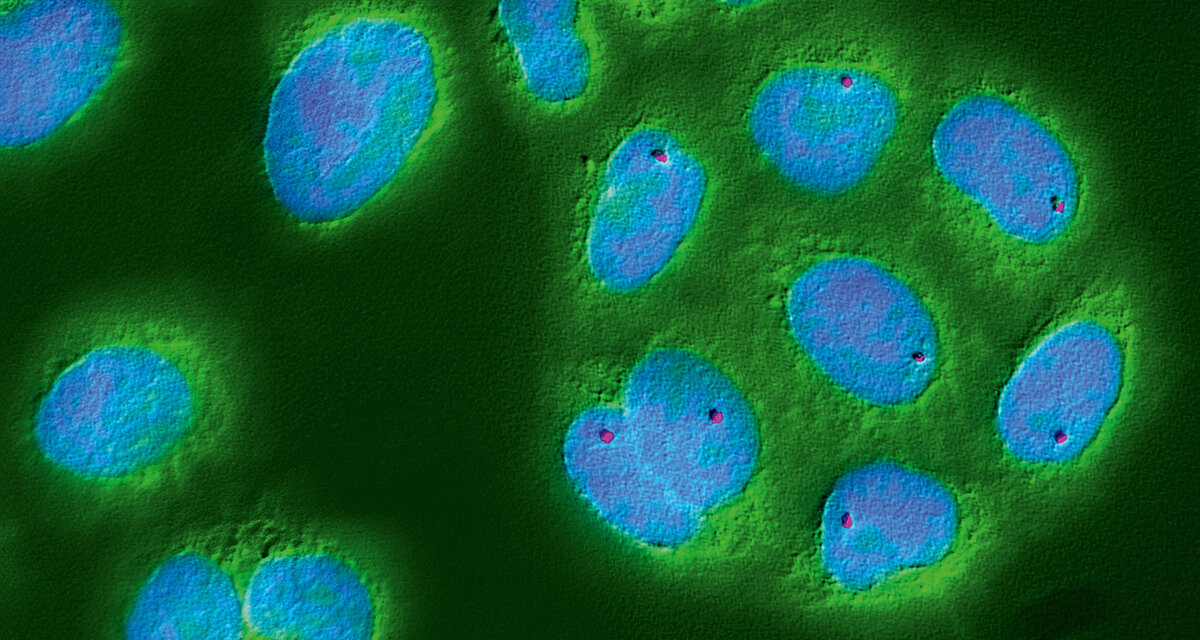
Wie wichtig ist FISH für die Leukämie-Befundung?
Nach wie vor sind Chromosomenanalysen und FISH verlässliche Routineverfahren für die Diagnose, Prognose und Verlaufskontrolle von Leukämien. Auch die WHO schreibt FISH-Diagnostik in ihren Guidelines vor. FISH beantwortet für Kliniker Fragen wie: Handelt es sich um Krebs oder eine andere hämatologische Systemerkrankung? Ist die akute Leukämie myeloid oder lymphoid? Zeigt der gefundenen zytogenetische Marker nach WHO-Kriterien eine günstige oder ungünstige Prognose an? War die Behandlung erfolgreich und wurden die Tumorzellen zurückgedrängt? Liegt ein Rezidiv mit denselben zytogenetischen Markern vor oder ist es eine sekundäre Leukämie mit komplett anderen chromosomalen Veränderungen? War die Knochenmarktransplantation erfolgreich? Weiterhin lässt sich bei akuten Leukämien mittels Zytogenetik und FISH der Ploidiegrad bestimmen.

Wann eignet sich FISH und wann Next Generation Sequencing, kurz NGS, besser?
Das hängt von der jeweiligen Fragestellung ab. FISH und Chromosomenanalysen erlauben Aussagen zu kleinen Zellmosaiken, die man mit NGS eher übersehen kann. Außerdem sind wir bei der FISH noch näher am In-vivo-Zustand. Im Vergleich zu NGS ist FISH schneller. In der hämatologischen Diagnostik kann NGS nur in Laboratorien mit sehr hohem Durchsatz wirtschaftlich eingesetzt werden. FISH können auch Einrichtungen mit kleineren Fallzahlen halbwegs kostendeckend anbieten. NGS erfasst gleichzeitig Dutzende bis Hunderte Mutationen in einer Probe, was die Interpretation erschwert. Beim Einsatz von FISH kann man nur eine Fragestellung bearbeiten und bei gleichzeitiger Durchführung der zytogenetischen Diagnostik auch ungerichtet nach grobstrukturellen Veränderungen suchen. Insofern können sich NGS und FISH auch gut ergänzen. Etwa um in Verlaufskontrollen einen mittels NGS gefundenen Tumormarker mit FISH weiter zu verfolgen.
Wird es eine Zukunft ohne FISH geben?
Es sollte nicht darum gehen, bewährte Techniken durch neue zu ersetzen, sondern darum, genau die Methode auszuwählen, die eine klinische Fragestellung bestmöglich klärt. FISH wird immer eine Rolle spielen.
Was sagen Sie zu der neuen IVDR-Zertifizierung, die bald verpflichtend sein soll?
Wenn ich die IVDR korrekt verstehe, darf man nur dann eine FISH-Sondenkombination in der Diagnostik einsetzen, wenn man genau diese zuvor an 100 Kontrollen und 100 auffälligen Patientenproben getestet hat. Das ist nicht mal für Labore oder Anbieter von Sonden, die eine Trisomie 21 nachweisen, möglich. Somit hieße das, weder in der Molekulargenetik noch in der FISH dürfte man die Diagnostik seltener Erkrankungen durchführen. Selbst hergestellte Sondensets wären nicht mehr legal. Da kein Labor den geforderten Aufwand erbringen kann, wäre man technisch wieder im Jahr 1981. Eine Untersuchung, wie auf Seite 56 zu sehen, wäre dann nicht mehr möglich. Wenn es keine gesetzlichen Last-minute-Änderungen gibt, dann kommt es dadurch zu einer deutlichen Verschlechterung der Patientenversorgung in ganz Europa.
INTERVIEW DR.JENDRIKMARBACH
HISTORIE DER ZYTOGENETIK
Einblicke in den Bauplan des Lebens
1879 | Flemming begründet die Zytogenetik |
| 1888 | Waldeyer-Hartz etabliert den Begriff „Chromosom“ für färbbare Strukturen im Zellkern, die Mendel als „Kopplungsgruppe“ postuliert |
1902/ 03 | Sutton und Boveri begründen die Chromosomen-Theorie der Vererbung |
1956 | Erkenntnis, dass Menschen 46 Chromosomen besitzen, und Entdeckung numerischer Aberrationen wie Trisomie 21 |
| 1960 | Nowell und Hungerford entdecken erste erworbene chromosomale Veränderung (Philadelphia-Chromosomen bei CML) |
| 1966 | erste pränatale Chromosomenanalyse |
| um 1970 | Einführung der Bänderungszytogenetik durch Lore Zech |
| 1981 | Entwicklung der ersten nicht-radioaktiven Sondenmarkierung mit Biotin als Hapten (nachweisbar durch Fluorchromgekoppeltes Avidin) |
| 1986 | erste Fluoreszenz-ISH (FISH) an menschlichen Chromosomen |
| 1989 | erste Vielfarben-FISH |
| 1996 | Meilenstein: gleichzeitige Verwendung aller 24 menschlichen Ganzchromosomenfarben in einem Experiment |
| Heute | FISH wird in der medizinischen Diagnostik und Forschung vielfältig eingesetzt |